Ich habe versucht, nach einer ähnlichen Frage zu suchen, konnte aber keine finden. Wenn Sie das tun, lassen Sie es mich bitte wissen! Getreide Heftklammern Blick aufWie ersetze ich eine NA durch einen äquivalenten Wert von einer anderen Stelle in einem Datensatz?
Hier ist eine Teilmenge von meinem Dataset
Ich habe an einem Projekt arbeiten:
nutrient.component. grain nutrients
1 Beta-carotene (μg) White Rice 0.00
2 Beta-carotene (μg) Brown Rice NA
3 Calcium (mg) White Rice 28.00
4 Calcium (mg) Brown Rice 23.00
5 Carbohydrates (g) White Rice 80.00
6 Carbohydrates (g) Brown Rice 77.00
7 Copper (mg) White Rice 0.22
8 Copper (mg) Brown Rice NA
9 Energy (kJ) White Rice 1528.00
10 Energy (kJ) Brown Rice 1549.00
11 Fat (g) White Rice 0.66
12 Fat (g) Brown Rice 2.92
13 Fiber (g) White Rice 1.30
14 Fiber (g) Brown Rice 3.50
15 Folate Total (B9) (μg) White Rice 8.00
16 Folate Total (B9) (μg) Brown Rice 20.00
17 Iron (mg) White Rice 0.80
18 Iron (mg) Brown Rice 1.47
19 Lutein+zeaxanthin (μg) White Rice 0.00
20 Lutein+zeaxanthin (μg) Brown Rice NA
21 Magnesium (mg) White Rice 25.00
22 Magnesium (mg) Brown Rice 143.00
23 Manganese (mg) White Rice 1.09
24 Manganese (mg) Brown Rice 3.74
25 Monounsaturated fatty acids (g) White Rice 0.21
26 Monounsaturated fatty acids (g) Brown Rice 1.05
27 Niacin (B3) (mg) White Rice 1.60
28 Niacin (B3) (mg) Brown Rice 5.09
29 Pantothenic acid (B5) (mg) White Rice 1.01
30 Pantothenic acid (B5) (mg) Brown Rice 1.49
31 Phosphorus (mg) White Rice 115.00
32 Phosphorus (mg) Brown Rice 333.00
33 Polyunsaturated fatty acids (g) White Rice 0.18
34 Polyunsaturated fatty acids (g) Brown Rice 1.04
35 Potassium (mg) White Rice 115.00
36 Potassium (mg) Brown Rice 223.00
37 Protein (g) White Rice 7.10
38 Protein (g) Brown Rice 7.90
39 Riboflavin (B2)(mg) White Rice 0.05
40 Riboflavin (B2)(mg) Brown Rice 0.09
41 Saturated fatty acids (g) White Rice 0.18
42 Saturated fatty acids (g) Brown Rice 0.58
43 Selenium (μg) White Rice 15.10
44 Selenium (μg) Brown Rice NA
45 Sodium (mg) White Rice 5.00
46 Sodium (mg) Brown Rice 7.00
47 Sugar (g) White Rice 0.12
48 Sugar (g) Brown Rice 0.85
49 Thiamin (B1)(mg) White Rice 0.07
50 Thiamin (B1)(mg) Brown Rice 0.40
51 Vitamin A (IU) White Rice 0.00
52 Vitamin A (IU) Brown Rice 0.00
53 Vitamin B6 (mg) White Rice 0.16
54 Vitamin B6 (mg) Brown Rice 0.51
55 Vitamin C (mg) White Rice 0.00
56 Vitamin C (mg) Brown Rice 0.00
57 Vitamin E, alpha-tocopherol (mg) White Rice 0.11
58 Vitamin E, alpha-tocopherol (mg) Brown Rice 0.59
59 Vitamin K1 (μg) White Rice 0.10
60 Vitamin K1 (μg) Brown Rice 1.90
61 Water (g) White Rice 12.00
62 Water (g) Brown Rice 10.00
63 Zinc (mg) White Rice 1.09
64 Zinc (mg) Brown Rice 2.02
Brauner Reis hat vier NA-Werte.
Basierend auf dieser Grafik, 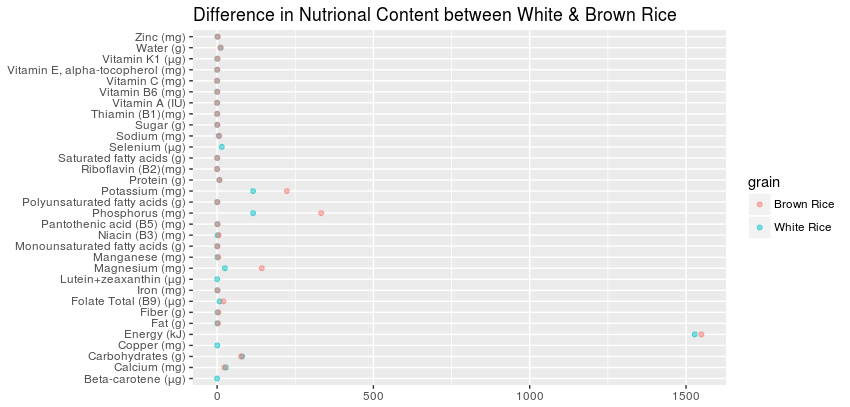 Ich denke, es wäre fair anzunehmen, dass die NA-Werte für braunen Reis werden sehr nahe an den entsprechenden Wert für weißen Reis. Und dass es genauer wäre, die Werte für weißen Reis zu spiegeln, anstatt die Werte auf Null zu setzen.
Ich denke, es wäre fair anzunehmen, dass die NA-Werte für braunen Reis werden sehr nahe an den entsprechenden Wert für weißen Reis. Und dass es genauer wäre, die Werte für weißen Reis zu spiegeln, anstatt die Werte auf Null zu setzen.
Meine Frage ist, neben der manuellen Suche und Eingabe des weißen Reisäquivalent Nährstoffes für braunen Reis, wie würde der Code aussehen, um NA durch den entsprechenden Wert für weißen Reis zu ersetzen? Ich würde erwarten, dass das Ergebnis den NA-Wert für Kupfer umrechnet; Brauner Reis, um den gleichen Wert wie Kupfer zu sein; Weißer Reis (der 0,22 ist). Wäre es besser, zuerst NA durch Null zu ersetzen? Aber wenn ich das tue, dann habe ich sechs Nährstoffe mit einem Wert von Null anstelle von vier Werten mit NA. Ich versuche, die richtige Einstellung zu finden, um dies durch Code zu lösen. Jeder Einblick in dieses würde sehr geschätzt werden.
Danke
Vielen Dank für diesen Tipp. Es scheint eine merkwürdig spezifische Funktion zu sein.Ich versuche mir vorzustellen, ob es andere Situationen gibt, in denen diese Funktion nützlich wäre? – RunAmuck
@RunAmuck Es ist üblich, dass Personen einen Wert für eine Zeile aufschreiben und die folgenden Zellen leer lassen, wenn ihre Werte gleich sind. Ich denke, dass die 'fill'-Funktion entworfen ist, um die" NA "oder leere Zellen in dieser Situation auszufüllen. – www