Ich habe etwas ähnliche Fragen gesehen, aber ich möchte meine konkrete Frage stellen so direkt wie ich kann:Transfarbskala, aber halten eine schöne Legende mit ggplot2
Ich habe ein Streudiagramm mit einem „z“ Variable in einer Farbskala kodiert:
library(ggplot2)
myData <- data.frame(x = rnorm(1000),
y = rnorm(1000))
myData$z <- with(myData, x * y)
badVersion <- ggplot(myData,
aes(x = x, y = y, colour = z))
badVersion <- badVersion + geom_point()
print(badVersion)
Welche dies erzeugt: 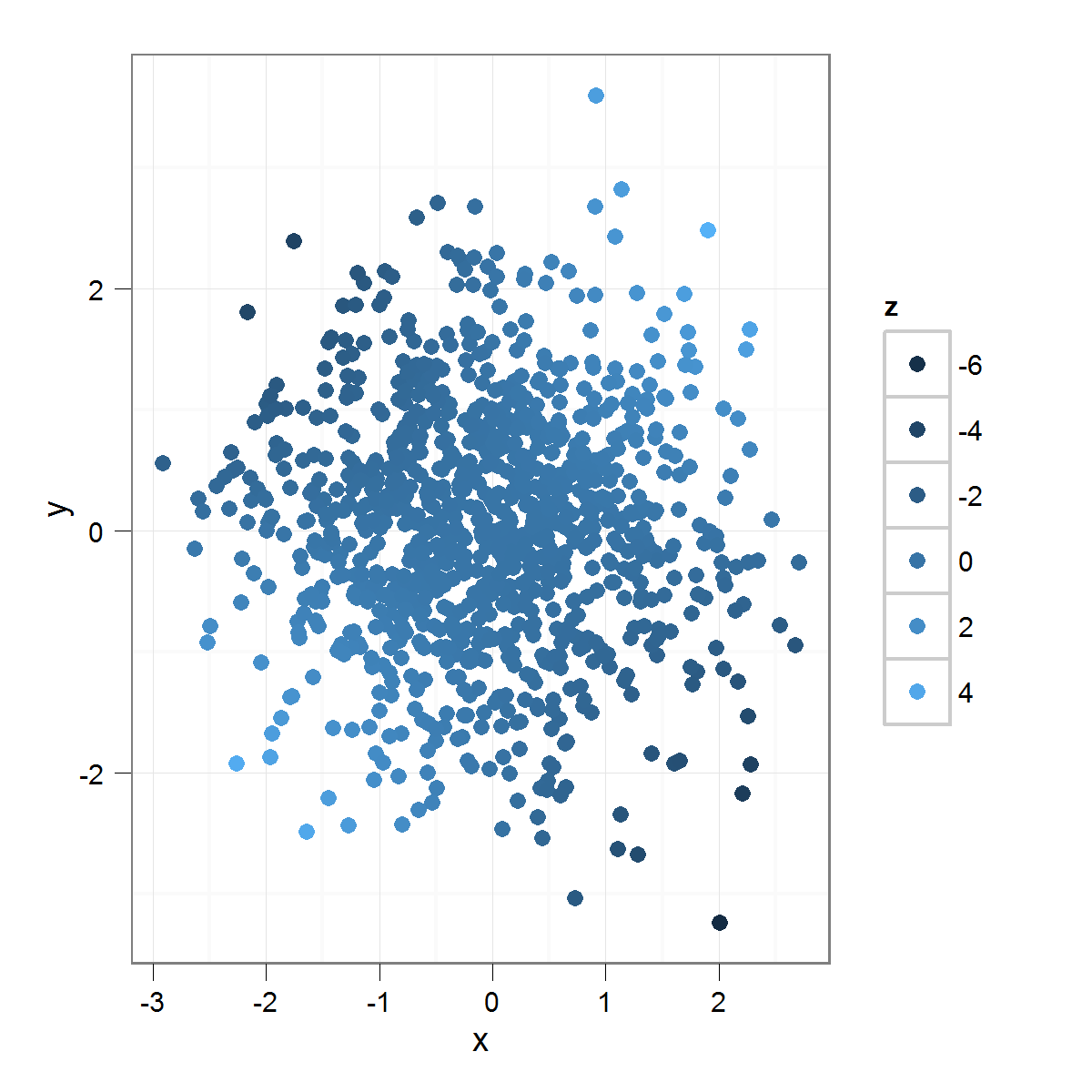
Wie Sie sehen können, da die „z“ Variable normalverteilt ist, nur sehr wenige der Punkte mit den sind farbig "extreme" Farben der Verteilung. Das ist so, wie es sein sollte, aber ich bin daran interessiert, Unterschiede zu betonen. Eine Möglichkeit, dies zu tun wäre, zu verwenden:
betterVersion <- ggplot(myData,
aes(x = x, y = y, colour = rank(z)))
betterVersion <- betterVersion + geom_point()
print(betterVersion)
Welche dies erzeugt: 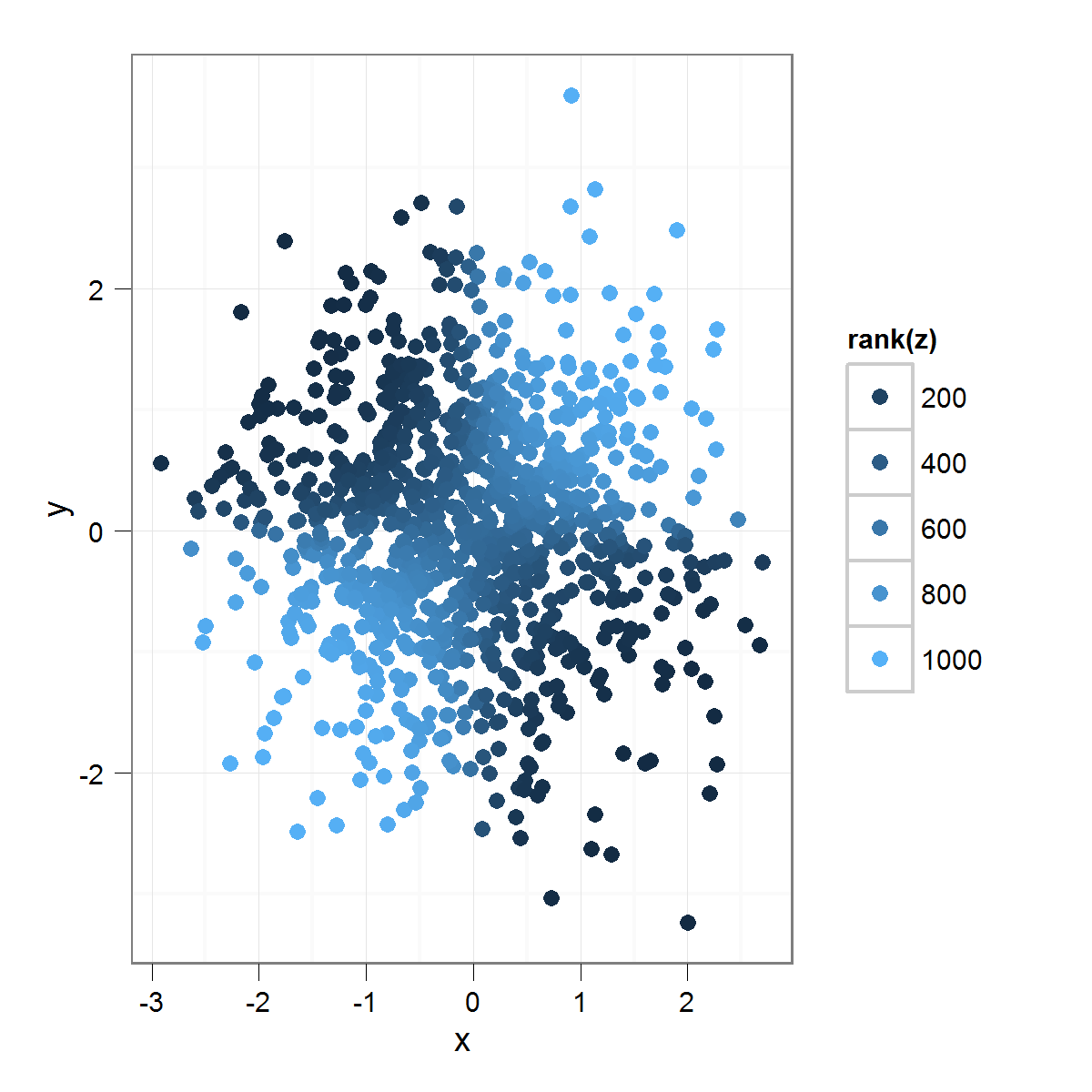
Durch die Anwendung Rang(), um die „z“ Variable, bekomme ich einen viel größeren Wert auf geringe Unterschiede innerhalb der " z "variabel. Man könnte sich vorstellen, hier irgendeine Transformation anstelle des Rangs zu verwenden, aber man bekommt die Idee.
Meine Frage ist im Wesentlichen, was ist der einfachste Weg, oder die "wahre ggplot2" Weg, eine Legende in den ursprünglichen Einheiten (Einheiten von z, im Gegensatz zu dem Rang von z) zu bekommen, während beibehalten die umgewandelte Version der farbigen Punkte?
Ich habe das Gefühl, dies verwendet rescaler() irgendwie, aber es ist mir nicht klar, wie rescaler() mit beliebigen Transformationen usw. zu verwenden ist. Im Allgemeinen wären klarere Beispiele nützlich.
Vielen Dank im Voraus für Ihre Zeit.


+1 für ein reproduzierbares Beispiel, klares Ziel und eine interessante Visualisierungsfrage. –